En el marco del proyecto DIMCoVAR, financiado con más de 200.000 euros por el Fondo Supera COVID de la CRUESantander, el Consejo Superior de Investigaciones Científicas (CSIC), a través del Instituto de Investigaciones Marinas (IIM), ha logrado la secuenciación del virus SARSCoV-2 en muestras de aguas residuales. “Desde que se detectó el material genético del virus en muestras de heces de pacientes infectados, ha habido un interés a nivel internacional por la monitorización del SARS-CoV-2 en aguas residuales”, destacan Antonio Figueras y Beatriz Novoa,
directores del grupo de Inmunología y Genómica del IIM.
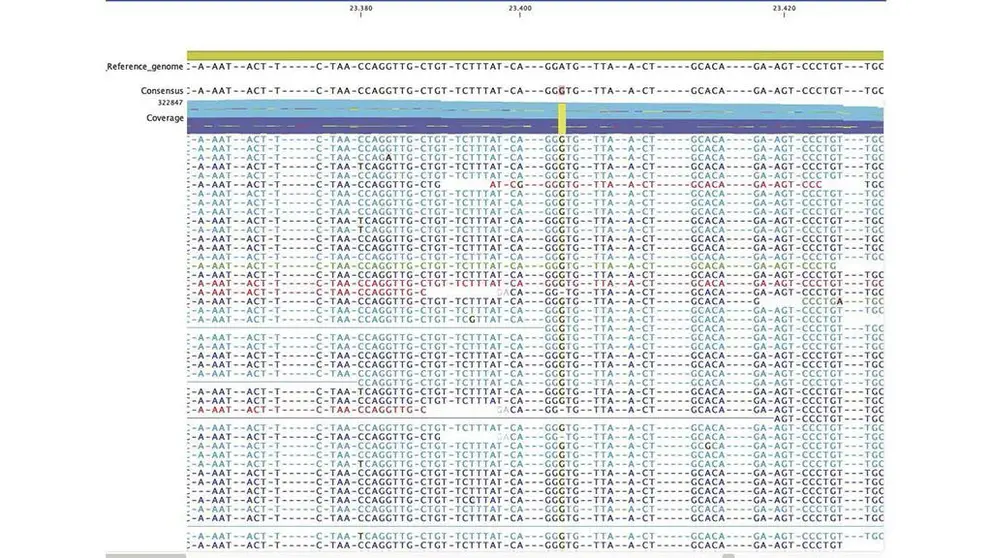
En un primer momento, el grupo de investigación intentó secuenciar las muestras de agua que habían dado resultados positivos mediante PCR con un elevado número de copias de genes virales, pero, “dado que el virus se halla muy fragmentado en las muestras de aguas residuales, la secuenciación no resulta fácil”, explican.
Por ello, desde el mes de septiembre, el grupo apostó por la aplicación de distintas metodologías para la secuenciación de las muestras. Ahora, aplicando técnicas de secuenciación masiva más sensibles y que requieren poca cantidad de material genético, ha obtenido millones de secuencias de SARS-CoV-2.
“Este logro es una enorme satisfacción para el grupo, pues desde hace un año estamos trabajando en esta línea. Primero, en el marco de un convenio con la Xunta de Galicia y con financiación del fondo Supera COVID. El conseguir detectar las mutaciones que están circulando en una localidad es muy valioso para identificar variantes. Hay que tener en cuenta que en las aguas residuales se concentra todo, incluso los virus de personas asintomáticas que no acuden al hospital. De ahí su importancia”, destaca Antonio Figueras.
El equipo de trabajo del CSIC, liderado por Antonio Figueras y Beatriz Novoa con la participación de Amaro Saco y Magalí Rey, ha logrado la secuenciación de muestras en tres localidades seleccionadas de entre las 15 objeto de seguimiento en el marco del proyecto DIMCoVAR: Baiona, Noia y Melide.
“En Noia y Melide las mutaciones detectadas son compatibles con la variante española y europea, mientras que en Baiona destacan las compatibles con la británica, sudafricana y brasileña. Dado que la mayoría de estas mutaciones se hallan en el gen que da lugar a la proteína Spike, que interactúa con los receptores celulares, se trata de mutaciones con implicaciones funcionales en el virus e interés clínico”, comenta Beatriz Novoa.
“Necesitamos secuenciar más, pero ya nos sorprende la cantidad de mutaciones que se encuentran en localidades como Baiona, con una prevalencia abrumadora de la variante británica, con mutaciones únicas que la definen; así como de mutaciones compartidas entre diversas variantes de preocupación entre las que se encuentran la
británica, sudafricana, brasileña y otras”, apunta Antonio Figueras.
El grupo de investigación también ha comparado estas secuencias con 500 genomas de SARS-Cov-2 secuenciados en pacientes clínicos gallegos. Esto ha derivado en la detección de mutaciones compartidas en las muestras de aguas residuales y pacientes de COVID-19, así como otras que no se habían detectado en personas afectadas por la enfermedad desde el inicio de la pandemia. Este trabajo permitiría la detección de nuevas mutaciones que pudiesen ser “peligrosas” de una forma más prematura.
“Todo ello reafirma la importancia de la monitorización de las aguas residuales para el seguimiento de las nuevas mutaciones del virus que puedan surgir”, concluyen desde el CSIC.
DIMCoVAR
Cuenta con la implicación, además del CSIC, de la Universidad de Vigo (UVigo) y la empresa Geseco Aguas. Abarca el análisis de la presencia de material genético de virus SARS- CoV-2 en las aguas residuales de 15 estaciones depuradoras de aguas residuales (EDAR) de Galicia, así como en los puntos de vertido de las EDAR en medio marino: Baiona, Nigrán, Gondomar, Cambados, Moraña, Puerto de Son, Muros, Melide, Ares, Cedeira, Noia, Pobra do Caramiñal, Betanzos, Viveiro y Burela.
La participación del CSIC, a través del IIM y, concretamente, de los grupos de investigación “Inmunología y Genómica” e “Ingeniería de Procesos” consiste fundamentalmente en la detección del material genético del virus por PCR y en el
desarrollo del modelo epidemiológico en base a los resultados de los análisis. La participación de la UVigo se corresponde al grupo de “BiotecnIA: Biotecnología Industrial e Ingeniería Ambiental”.